
 Autour du thème des TIPE pour les concours 2020
Exposé 1
Exposé 2
Exposé 3
Exposé 4
Autour du thème des TIPE pour les concours 2020
Exposé 1
Exposé 2
Exposé 3
Exposé 4
 Autres années
Autour du thème des TIPE pour les concours 2019 - Transports
Autres années
Autour du thème des TIPE pour les concours 2019 - Transports

 Autour du thème des TIPE pour les concours 2020
Exposé 1
Exposé 2
Exposé 3
Exposé 4
Autour du thème des TIPE pour les concours 2020
Exposé 1
Exposé 2
Exposé 3
Exposé 4
 Autres années
Autour du thème des TIPE pour les concours 2019 - Transports
Autres années
Autour du thème des TIPE pour les concours 2019 - Transports
Des vitesses record rendent la spectroscopie Raman spontanée plus pratique pour les applications biomédicales.
La spectroscopie Raman est une technique non invasive bien connue pour déterminer la composition chimique d’échantillons complexes. Par exemple, elle s’est révélée prometteuse pour l’identification des cellules cancéreuses et l’analyse de tissus à la recherche de maladies. Bien que cette méthode soit remarquablement simple (aucune préparation d’échantillon n’est nécessaire), elle nécessite généralement des vitesses d’acquisition d’images trop lentes pour saisir la dynamique des échantillons biologiques. De plus, le traitement de l’énorme quantité de données générées par l’imagerie spectroscopique prend beaucoup de temps et constitue souvent une limite à l’étude de spécimens de grande surface.
Dans le cadre d’une collaboration internationale à laquelle participent le LPENS et le LKB, une méthodologie a été mise au point pour relever simultanément deux défis : augmenter la vitesse d’échantillonnage et introduire un moyen plus simple d’obtenir des informations utiles des images spectroscopiques. Ces travaux ont été publiés dans la revue open-source Optica.
Pour accélérer le processus d’imagerie, les chercheurs ont modifié leur système Raman en remplaçant les caméras lentes et coûteuses utilisées dans les installations conventionnelles par un modulateur spatial de lumière rapide et bon marché, aussi appelé « matrice de micro-miroirs ». Cet appareil sélectionne des groupes de longueurs d’onde qui sont détectées par un détecteur à pixel unique très sensible. Ils n’acquièrent délibérément pas l’ensemble des données nécessaires à l’imagerie spectroscopique Raman classique, comprimant les images au fur et à mesure qu’elles sont acquises. Côté logiciel, ils complètent les informations manquantes en exploitant un algorithme très spécial : il s’agit en fait d’un algorithme reconfiguré, développé à l’origine pour la compétition de prédiction des préférences cinématographiques 2009 de Netflix !
Les chercheurs ont testé leur nouvelle méthodologie d’imagerie sur des tissus cérébraux et des cellules individuelles, qui présentent tous deux une grande complexité chimique. Leurs résultats ont montré que la méthode permet d’acquérir des images à des vitesses de quelques dizaines de secondes, alors que le Raman traditionnel prenait généralement quelques minutes. Ils compriment en même temps les données acquises - en réduisant leur volume d’un facteur 64 !
Les chercheurs pensent que cette nouvelle approche devrait fonctionner avec la plupart des échantillons biologiques, mais ils prévoient la tester avec d’autres types de tissus pour le démontrer expérimentalement. En plus des outils cliniques, la méthode pourrait être utile pour des applications biologiques telles que la caractérisation des algues. Ils veulent également améliorer la vitesse de balayage de leur système afin de réaliser une acquisition d’images en moins d’une seconde. Une telle avancée pourrait rendre la méthode d’imagerie Raman utilisable pour des applications cliniques telles que la détection de tumeurs ou l’analyse tissulaire.
Hilton B. de Aguiar, coordinateur de la collaboration, est titulaire d’une Junior Research Chair au département de physique de l’ENS.
Original Paper : F. Soldevila, J. Dong, E. Tajahuerce, S. Gigan, H. B. De Aguiar, “Fast compressive Raman bio-imaging via matrix completion,” Optica 6, 341-346 (2019).
DOI : https://doi.org/10.1364/OPTICA.6.000341
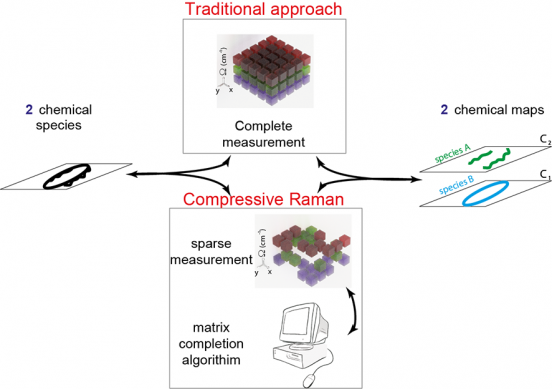
Figure 1 Imagerie Raman Compressive - Avec cette nouvelle approche, lil est possible d’acquérir moins de données spectrales que ce qui est traditionnellement requis, puis utiliser un algorithme de complétion pour compléter les informations non enregistrées.
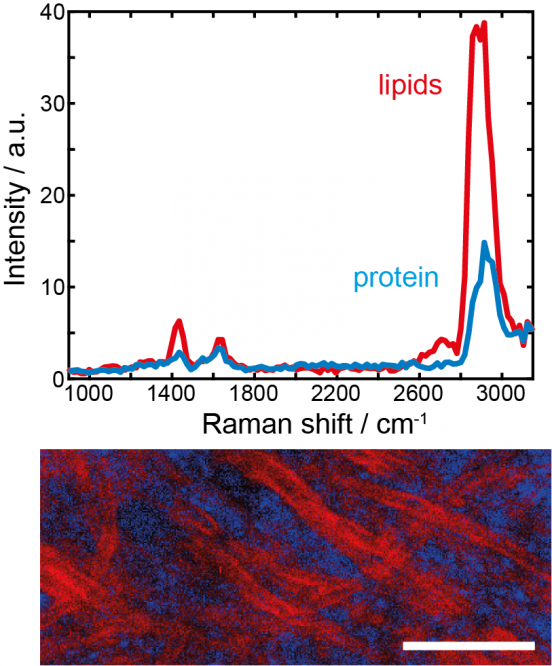
Figure 2 Spectroscopie Raman d’un tissu cérébral --- Barre d’échelle : 20 microns.
Credit : Hilton De Aguiar, École Normale SupérieureAuteur correspondant :
Contact communication :
 RECRUTEMENT
RECRUTEMENT ACCES DIRECT
ACCES DIRECT